Vue d'ensemble
Pipeline local de data engineering pour données hospitalières (Diabetes 130) avec ingestion, transformation et orchestration via Airflow, MinIO, PostgreSQL et Python.
Ce projet a été conçu pour simuler un environnement de traitement de données en local, en reproduisant des pratiques courantes du data engineering dans l’industrie. Le jeu de données utilisé (Diabetes 130‑US Hospitals) contient plus de 100 000 admissions hospitalières de patients atteints de diabète, avec des variables liées aux diagnostics, traitements et séjours.
L’objectif était de construire un pipeline complet permettant :
- l’ingestion des données sources dans un stockage objet local (MinIO),
- leur transformation avec Python et Pandas (nettoyage, filtrage, enrichissement),
- leur chargement dans une base PostgreSQL pour des usages analytiques,
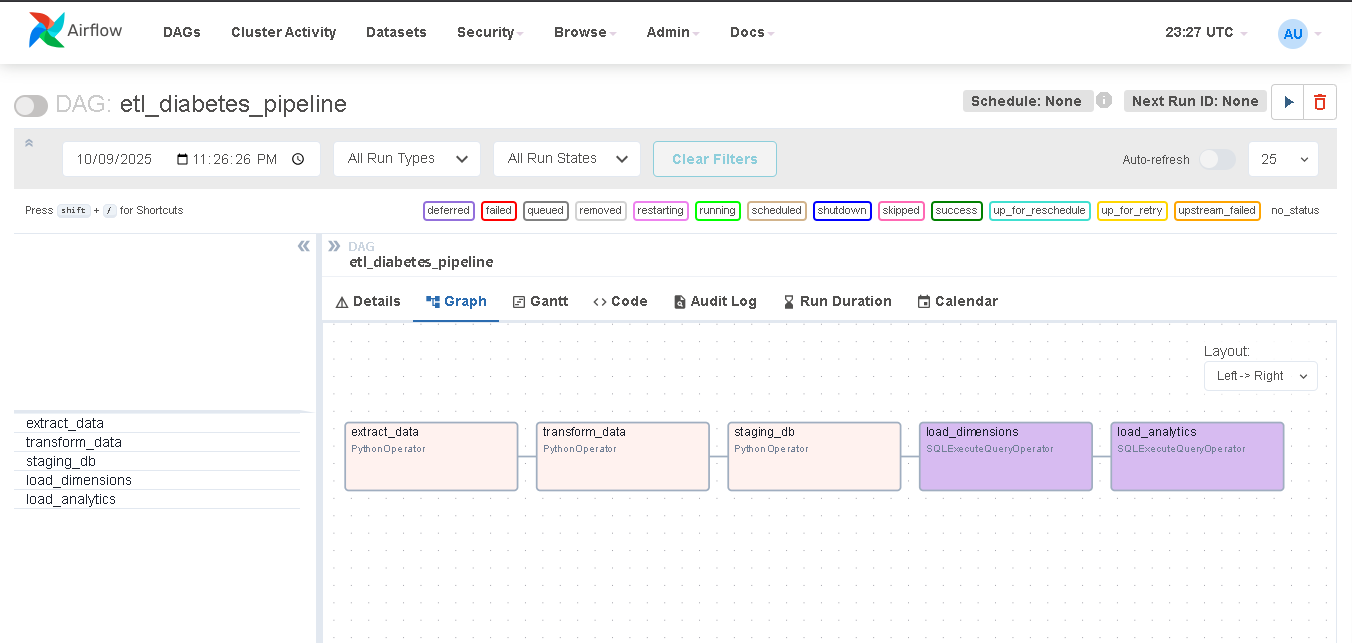
- l’orchestration des tâches avec Apache Airflow (DAGs structurés),
- le suivi de l’état du pipeline via des logs et du monitoring.
Ce projet met en œuvre les principes fondamentaux de l’ETL (Extract, Transform, Load) et démontre la capacité à construire un flux de traitement de données reproductible, traçable et maintenable, adapté à un contexte data analytique ou santé.
Galerie
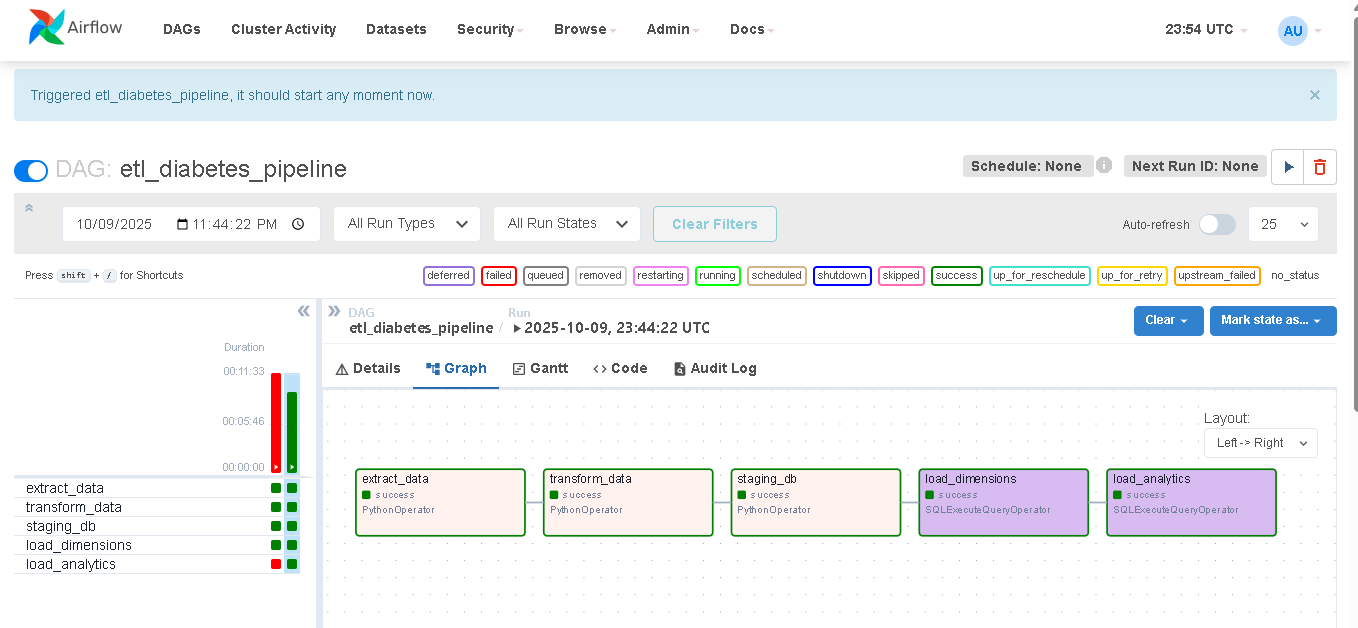
Pipeline Airflow
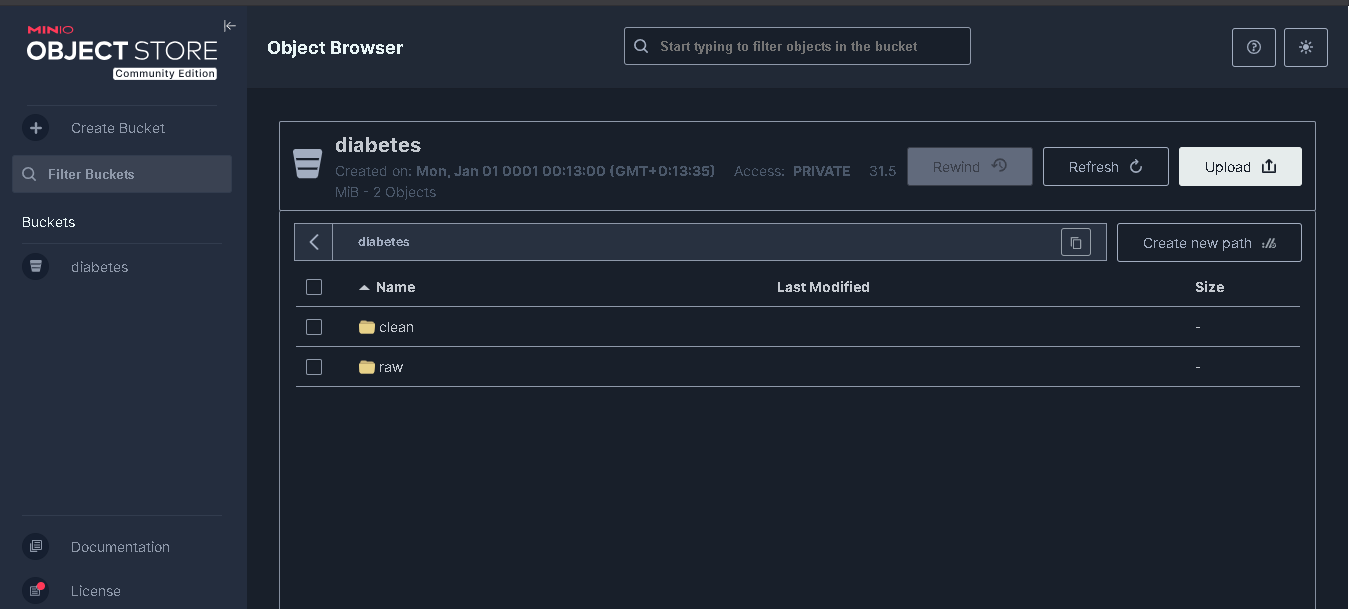
Minio Dashboard
Tech Stack
PostgreSQLApache AirflowMinio S3PythonDocker
Infos Projet
TypeIndividuel
Statuscompleted